آموزش PyG (pytorch-geometric) برای کار با داده های گرافی
امروزه پیشرفتهای روز افزون هوش مصنوعی در زمینه های مختلف بر هیچ کس پوشیده نیست. در دنیای هوش مصنوعی با با انواع مختلفی از داده ها نظیر تصویر، متن، صوت و یا داده های ساختار یافته مواجه هستیم. اما یکی از انواع داده که در هیچ کدام از این دسته ها جای نمیگیرد داده های گرافی است که اخیرا پیشرفت های زیادی را در هوش مصنوعی رقم شده است. برای مثال ما میتوانیم مولکولهای دارو را به صورت گرافی از اتم ها و پیوندها تعریف کنیم و یک آنتی بیوتیک جدید کشف کنیم! فریم ورکهای زیادی برای کار با داده ها گرافی وجود دارد، در اینجا مفاهیم پایه پایتورج جئومتریک برای پیاده سازی شبکه های عصبی گرافی را بررسی خواهیم کرد.
مفاهیم پایهی PyG (pytorch-geometric) برای کار با داده های گراف
ما به طور کوتاه مفاهیم اساسی PyG را در این پست بررسی میکنیم. برای آشنایی با مفاهیم Graph Machine Learning، میتوانید از کورس انگلیسی Stanford CS22W: Machine Learning with Graphs یا دورهی آموزش فارسی شبکههای عصبی گرافی استفاده کنید. البته منابع مرتبط با شبکه های عصبی گرافی را نیز میتوانید مطالعه کنید.
نکته:
- در این مقاله کتابخانهی pytorch geometric و PyG و پایتورچ جئومتریک با یک مفهوم یکسان که به این کتابخانه اشاره دارد استفاده شده است.
- هر جا از واژهی گره یا راس استفاده کرده ایم منظور Node در یک گراف است.
- و هر جا از یال استفاده کردهایم منظور Edge در گراف بوده است.
مدیریت داده های گرافها
یک گراف برای مدل سازی روابط زوجی (یالها) بین اشیا (رئوس) استفاده می شود. یک گراف منفرد در PyG با نمونه ای از torch_geometric.data.Data توصیف می شود که ویژگی های زیر را به طور پیش فرض نگه می دارد:
data.x: ماتریس ویژگیهای رئوس با شکل [num_nodes, num_node_features]
data.edge_index: اتصالات گراف در قالب COO با شکل [2, num_edges] و تایپ torch.long
data.edge_attr: ماتریس ویژگیهای یال با شکل [num_edges, num_edge_features]
data.y: لیبل برای آموزش (ممکن است شکل دلخواه داشته باشد)، به عنوان مثال، مثلا برای طبقه بندی رئوس شکل [تعداد_گره ها، *] یا طبقه بندی سطح گراف شکل [1، *]
data.pos: ماتریس موقعیت گره با شکل [num_nodes, num_dimensions]
نمونه کد:
import torch
from torch_geometric.data import Data
edge_index = torch.tensor([[0, 1, 1, 2],
[1, 0, 2, 1]], dtype=torch.long)
x = torch.tensor([[-1], [0], [1]], dtype=torch.float)
data = Data(x=x, edge_index=edge_index)
>>> Data(edge_index=[2, 4], x=[3, 1])
توجه کنید که `edge_index`، به عبارتی تانسوری است که lمبدا و مقصد تمامی یالها را تعریف میکند ولی نباید آن را به عنوان یک لیست از جفت اعداد در نظر بگیرید. اگر شما میخواهید شاخصهای خود را به این صورت تعریف کنید (یعنی ماتریس n در 2 به جای ماتریس 2 در n)، باید قبل از فراخوانی Data آن را ترانهاده (transpose) کرده و سپس با فراخوانی تابع contiguous آنها را به فرمت مورد نظر فریم ورک تبدیل کنید.
import torch
from torch_geometric.data import Data
edge_index = torch.tensor([[0, 1],
[1, 0],
[1, 2],
[2, 1]], dtype=torch.long)
x = torch.tensor([[-1], [0], [1]], dtype=torch.float)
data = Data(x=x, edge_index=edge_index.t().contiguous())
>>> Data(edge_index=[2, 4], x=[3, 1])
همچنین در مثال بالا اگرچه گراف فقط دو یال دارد، اما باید چهار تاپل شاخص تعریف کنیم تا هر دو جهت یک یال را محاسبه کنیم.
توجه کنید که ضروری است که عناصر edge_index فقط شامل اعدادی در بازه { 0، …، num_nodes – 1} باشند. این الزامی است زیرا ما می خواهیم نمایش داده نهایی خود را به صورت جمع و جوری داشته باشیم، به عنوان مثال، ما می خواهیم ویژگی های راس مبدا و مقصد یال اول (0، 1) را از طریق x [0] و x [1] به ترتیب فراخوانی کنیم. شما همیشه می توانید با اجرای validate() بررسی کنید که شیء داده نهایی خود این الزامات را ارضا می کند یا خیر.
data.validate(raise_on_error=True)
شئ Data در این فریم ورک علاوه بر داشتن خصیصه هایی مرتبط با سطح گره، سطح لبه یا سطح گراف، تعدادی توابع کاربردی مفید را نیز ارائه می دهند، به عنوان مثال:
print(data.keys)
>>> ['x', 'edge_index']
print(data['x'])
>>> tensor([[-1.0],
[0.0],
[1.0]])
for key, item in data:
print(f'{key} found in data')
>>> x found in data
>>> edge_index found in data
'edge_attr' in data
>>> False
data.num_nodes
>>> 3
data.num_edges
>>> 4
data.num_node_features
>>> 1
data.has_isolated_nodes()
>>> False
data.has_self_loops()
>>> False
data.is_directed()
>>> False
# Transfer data object to GPU.
device = torch.device('cuda')
data = data.to(device)
شما می توانید لیست کاملی از همه متدها را در مستندات torch_geometric.data.Data پیدا کنید.
مجموعهدادههای رایج گرافی برای بنچمارک گیری
پایتورچ جئومتریک حاوی تعداد زیادی از مجموعه دادههای رایج بنچمارک مانند همه مجموعه دادههای Planetoid (کورا، سیتیسیر، پابمد)، همه مجموعه دادههای دسته بندی گراف از http://graphkernels.cs.tu-dortmund.de و نسخههای پیش پردازش شده آنها، مجموعه دادههای QM7 و QM9 و تعداد کمی از مجموعه دادههای سه بعدی شبکه مثل فائوست، مدلنت۱۰/۴۰ و شیپنت وجود دارد.
شروع به کار با مجموعه دادهها بسیار ساده است. هنگامی که یک مجموعه داده را شروع به اولین بار میکنید، فایلهای خام آن به صورت خودکار دانلود میشوند و به فرمت دادههای قبلاً توصیف شده در PyG پردازش میشوند. به عنوان مثال، برای بارگیری مجموعه داده ENZYMES (شامل ۶۰۰ گراف در ۶ کلاس)، با نوشتن دستور زیر این عمل انجام میشود:
from torch_geometric.datasets import TUDataset dataset = TUDataset(root='/tmp/ENZYMES', name='ENZYMES') >>> ENZYMES(600) len(dataset) >>> 600 dataset.num_classes >>> 6 dataset.num_node_features >>> 3
اکنون به تمام 600 گراف موجود در مجموعه داده دسترسی داریم:
data = dataset[0] >>> Data(edge_index=[2, 168], x=[37, 3], y=[1]) data.is_undirected() >>> True
میتوانیم ببینیم که گراف اول در مجموعه داده شامل ۳۷ گره است، هرکدام با سه ویژگی.
این گراف ۸۴ یال بدون جهت وجود دارد (168/2 = 84)
و هر گراف در این مجموعه داده به یک کلاس تعلق میگیرد. علاوه بر این مشخص است که، شیء data یک مساله graph-level یا سطح-گراف است.
همچنین با اندیس دهی مییتوانیم این دیتاست را تقسیم کنیم، مثال زیر را مشاهده کنید:
train_dataset = dataset[:540] >>> ENZYMES(540) test_dataset = dataset[540:] >>> ENZYMES(60)
که در این مثال 540 گراف را برای آموزش و 60 گراف را برای تست یا ارزابی قرار دادیم.
همچنین اگر مطمئن نیستید که آیا مجموعه داده قبلاً قبل از این تقسیم بندی (train/test) شافل شده یا به هم ریخته شده است یا خیر، میتوانید با این کد این کار را انجام دهید:
dataset = dataset.shuffle() >>> ENZYMES(600)
حالا بیاید نگاهی به دیتاست Cora بیندازیم:
from torch_geometric.datasets import Planetoid dataset = Planetoid(root='/tmp/Cora', name='Cora') >>> Cora() len(dataset) >>> 1 dataset.num_classes >>> 7 dataset.num_node_features >>> 1433
در اینجا، مجموعه داده فقط شامل یک گراف منفرد و بدون جهت است:
data = dataset[0] >>> Data(edge_index=[2, 10556], test_mask=[2708], train_mask=[2708], val_mask=[2708], x=[2708, 1433], y=[2708]) data.is_undirected() >>> True data.train_mask.sum().item() >>> 140 data.val_mask.sum().item() >>> 500 data.test_mask.sum().item() >>> 1000
این بار، شیء Data حاوی برچسبی (Label) برای هر گره بوده و خصیصههای بیشتری در سطح گره دارد که عبارتند از: train_mask، val_mask و test_mask، که هر کدام به شرح زیر هستند:
- train_mask نشان می دهد که در چه گره هایی باید در فرایند train یا آموزش شرکت داده شوند (۱۴۰ گره)
- val_mask نشان می دهد که کدام گره ها برای اعتبار سنجی و استفاده در early stopping مناسب هستند (۵۰۰ گره)
- test_mask نشان می دهد که چه گره هایی باید در فرایند تست و ارزیابی نهایی استفاده شوند (۱۰۰۰ گره)
مینیبچ (Mini-batche) در گراف
شبکههای عصبی معمولا به صورت دستهدسته یا همان هر دفعه یک batch آموزش داده میشوند. PyG موازی سازی در هر بچ را با ساختار ماتریسهای همجواری اسپارسی که توسط edge_index تعریف شدهاند و ادغام ماتریسهای ویژگی و هدف در بعد گره به دست می آورد. این ترکیب تعداد متفاوتی از گره ها و لبه ها را بر روی نمونه ها در یک batch را امکان پذیر می کند:
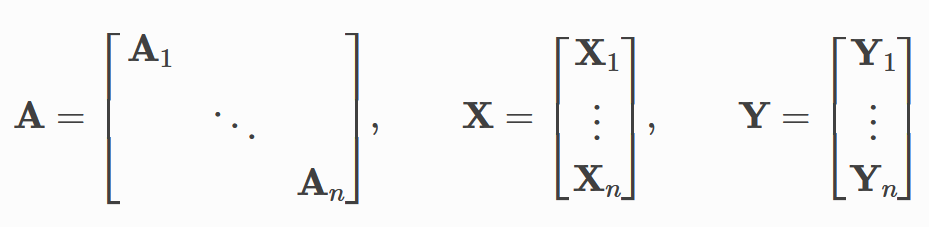
PyG شامل DataLoader خود به نام torch_geometric.loader است که برای پیوند دادن داده ها مراقبت استفاده می کند.
بیایید با مثالی درک کنیم:
from torch_geometric.datasets import TUDataset
from torch_geometric.loader import DataLoader
dataset = TUDataset(root='/tmp/ENZYMES', name='ENZYMES', use_node_attr=True)
loader = DataLoader(dataset, batch_size=32, shuffle=True)
for batch in loader:
batch
>>> DataBatch(batch=[1082], edge_index=[2, 4066], x=[1082, 21], y=[32])
batch.num_graphs
>>> 32
در واقع torch_geometric.data.Batch از کلاس torch_geometric.data.Data به ارث برده شده است و دارای خصیصهای اضافی به نام batch است.
batch یک بردار ستونی است که هر گره یا راس را به گراف مربوطه خود در نگاشت میکند.
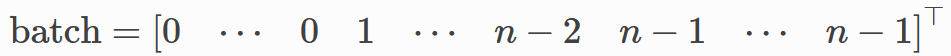
این ویژگی میتواند برای محاسبه میانگین ویژگیهای گره در بعد گره برای هر گراف به صورت جداگانه استفاده شود.
from torch_geometric.utils import scatter
from torch_geometric.datasets import TUDataset
from torch_geometric.loader import DataLoader
dataset = TUDataset(root='/tmp/ENZYMES', name='ENZYMES', use_node_attr=True)
loader = DataLoader(dataset, batch_size=32, shuffle=True)
for data in loader:
data
>>> DataBatch(batch=[1082], edge_index=[2, 4066], x=[1082, 21], y=[32])
data.num_graphs
>>> 32
x = scatter(data.x, data.batch, dim=0, reduce='mean')
x.size()
>>> torch.Size([32, 21])
کد بالا در ابتدا اطلاعات گرافی مربوط به پروتئینها را بارگیری میکند. در اینجا، دیتاست پروتئین ENZYMES استفاده شده است. این کد برای هر دسته از دادههای آموزشی 32 تایی، میانگین ویژگی هر گره را به دست میآورد.
در این کد، ابتدا با استفاده از تابع TUDataset دیتاست ENZYMES از مسیر ‘/tmp/ENZYMES’ بارگیری شده و در متغیر dataset ذخیره میشود. سپس با استفاده از DataLoader، دادههای این دیتاست به صورت دستهای با اندازه 32 تایی خوانده میشوند و در متغیر loader قرار گرفته و در هر تکرار حلقه for، دادههای هر دسته به صورت یک DataBatch به متغیر data اختصاص داده میشوند.
DataBatch شامل سه ورودی است:
– batch، که یک لیست از طول تعداد گرههای هر گراف در هر دسته است.
– edge_index، که شامل لیست یالهای هر گراف در هر دسته است.
– x، که ماتریس ویژگیهای هر گراف در هر دسته است.
در خط بعدی، تعداد گرافهای هر دسته با فراخوانی متد num_graphs بر روی متغیر data به دست میآید.
سپس با استفاده از تابع scatter با ورودیهای data.x (ماتریس ویژگیها) و data.batch (لیست طول گرههای هر گراف) به دستهبندی بر اساس بچ و محاسبه میانگین، ماتریس x به دست میآید. در نهایت، اندازه ماتریس x با فراخوانی تابع size() چاپ میشود.
تبدیل دادهها
اگر قبلا با فریم ورک پایتورچ برای کاربردهای بینایی کامپیوتر کار کرده باشید، میدانید که تبدیلها یک روش معمول در torchvision برای تبدیل تصاویر و انجام کارهایی نظیر augmentation داده هستند. PyG نیز تبدیلهای خاص کاربردهای خودش را دارد که ورودی آنها یک شیء Data است و یک شیء Dataجدید که تبدیلاتی روی آن اعمال شده را به عنوان خروجی برمیگرداند. تبدیلها میتوانند با استفاده از تابع Compose موجود در torch_geometric.transforms.Compose به هم پیوند داده شوند و قبل از ذخیره کردن مجموعه دادههای پردازش شده در دیسک (pre_transform) و یا قبل از دسترسی به یک گراف در مجموعه داده (transform) اعمال میشوند.
بیایید به مثالی نگاه کنیم که در آن تبدیلها را روی مجموعه داده ShapeNet (شامل ۱۷۰۰۰ پوینت کلاود سهبعدی و برچسب per point از ۱۶ دستهی شکل) اعمال میکنیم.
from torch_geometric.datasets import ShapeNet dataset = ShapeNet(root='/tmp/ShapeNet', categories=['Airplane']) dataset[0] >>> Data(pos=[2518, 3], y=[2518])
ما میتوانیم مجموعه دادههای point cloud را با ایجاد نزدیکترین همسایه از این point cloud و گراف نزدیکترین همسایه ها از طریق تبدیلها به یک مجموعه داده گراف تبدیل کنیم:
import torch_geometric.transforms as T
from torch_geometric.datasets import ShapeNet
dataset = ShapeNet(root='/tmp/ShapeNet', categories=['Airplane'],
pre_transform=T.KNNGraph(k=6))
dataset[0]
>>> Data(edge_index=[2, 15108], pos=[2518, 3], y=[2518])
ما از تابع pre_transform برای تبدیل دادهها قبل از ذخیره آنها در دیسک استفاده میکنیم (که منجر به زمان بارگذاری سریعتر میشود). لازم به ذکر است که در بارگیری بعدی مجموعه دادهها، حتی اگر شما هیچ تبدیلی را انجام ندهید ، دیتاست حاوی یالهای گراف خواهد بود. اگر pre_transform با مجموعه دادههای قبلاً پردازش شده همخوانی نداشته باشد نیز هشداری دریافت خواهید کرد.
علاوه بر این، ما می توانیم با استفاده از پارامتر transform ، یک شیء Data را به صورت تصادفی افزایش دهیم. به عنوان مثال، می توانیم هر موقعیت گره را با یک عدد کوچک ترجمه کنیم.
import torch_geometric.transforms as T
from torch_geometric.datasets import ShapeNet
dataset = ShapeNet(root='/tmp/ShapeNet', categories=['Airplane'],
pre_transform=T.KNNGraph(k=6),
transform=T.RandomJitter(0.01))
dataset[0]
>>> Data(edge_index=[2, 15108], pos=[2518, 3], y=[2518])
شما می توانید لیست کاملی از تمام تبدیل های پیاده سازی شده برای گراف را در torch_geometric.transforms پیدا کنید.
روشهای یادگیری روی گراف
پس از آشنایی با روش های برخورد با دادههای گرافی، مجموعه داده های موجود، اشیاء Data loader و تبدیلات مختلف، زمان پیاده سازی اولین شبکه عصبی گرافی فرا رسیده!
در اینجا از یک لایه ساده GCN استفاده خواهیم کرد و آزمایشات روی مجموعه داده های Cora انجام خواهد شد. برای توضیحات بیشتر درباره GCN میتوانید به دوره آموزشی شبکه های عصبی گرافی مراجع کنید.
اولین قدمی که نیاز داریم، لود کردن مجموعه داده Cora است.
from torch_geometric.datasets import Planetoid dataset = Planetoid(root='/tmp/Cora', name='Cora') >>> Cora()
توجه داشته باشید که در این مثال ما نیازی به استفاده از تبدیل یا دیتالودر نداریم. حالا بیایید یک GCN دو لایه را پیاده سازی کنیم:
import torch
import torch.nn.functional as F
from torch_geometric.nn import GCNConv
class GCN(torch.nn.Module):
def __init__(self):
super().__init__()
self.conv1 = GCNConv(dataset.num_node_features, 16)
self.conv2 = GCNConv(16, dataset.num_classes)
def forward(self, data):
x, edge_index = data.x, data.edge_index
x = self.conv1(x, edge_index)
x = F.relu(x)
x = F.dropout(x, training=self.training)
x = self.conv2(x, edge_index)
return F.log_softmax(x, dim=1)
کد بالا یک شبکه عصبی با دو لایه GCNConv را که در forward pass شبکه فراخوانی میشود، پیاده سازی میکند. توجه کنید که غیرخطی بودن در فراخوانی های conv در نظر گرفته نشده است و به همین دلیل باید پس از آن اعمال شود (چیزی که در تمام اپراتورهای PyG پایدار است). در اینجا، از ReLU را به عنوان واحد غیرخطی میانی استفاده شده و در نهایت softmax در خروجی قرار داده شده تا یک مساله طبقه بندی را حل کنیم. بیایید این مدل را بر روی ۲۰۰ epoch از نودهای آموزشی آموزش دهیم.
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
model = GCN().to(device)
data = dataset[0].to(device)
optimizer = torch.optim.Adam(model.parameters(), lr=0.01, weight_decay=5e-4)
model.train()
for epoch in range(200):
optimizer.zero_grad()
out = model(data)
loss = F.nll_loss(out[data.train_mask], data.y[data.train_mask])
loss.backward()
optimizer.step()
در نهایت مدل آموزش دیده را میتوانیم روی مجموعه رئوس تست ارزیابی کنیم:
model.eval()
pred = model(data).argmax(dim=1)
correct = (pred[data.test_mask] == data.y[data.test_mask]).sum()
acc = int(correct) / int(data.test_mask.sum())
print(f'Accuracy: {acc:.4f}')
>>> Accuracy: 0.8150
این مستندات، توابع پایه برای کار با داده های گرافی و شبکه های عصبی گرافی را فراهم میکنند.




دیدگاهتان را بنویسید