مجموعه دادهی PPI (Protein-Protein Interaction)
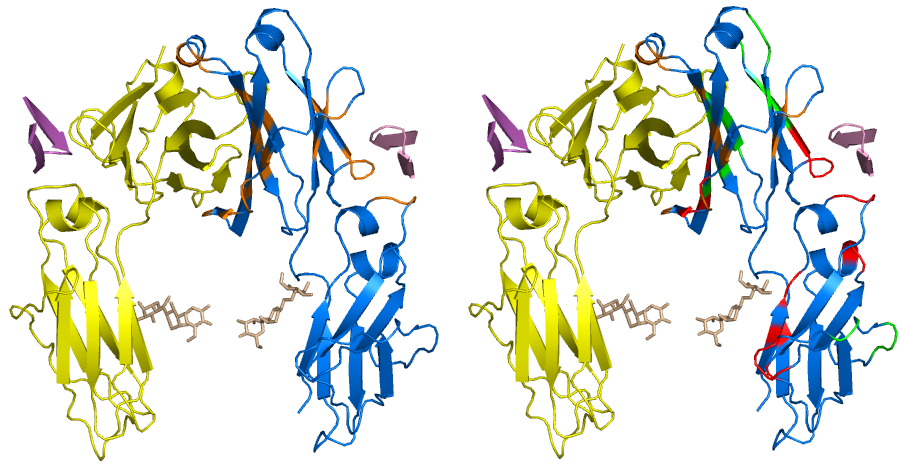
در PyTorch Geometric، مجموعه دادهی PPI (Protein-Protein Interaction) یک مجموعه داده بنچمارک برای کارهای طبقهبندی گراف است. این مجموعه شامل گرافهایی است که نشاندهنده تعاملات پروتئین ها هستند و در آن هر گره یک پروتئین را نشان میدهد و هر یال نشان دهنده تعامل بین دو پروتئین است. هدف از این مجموعهداده، پیشبینی این است که آیا شبکه تعامل پروتئین-پروتئینی مورد نظر با یک تعامل مثبت یا منفی پروتئین-پروتئین مطابقت دارد.
این مجموعه دادهی تعامل پروتئین-پروتئین در مقاله “پیش بینی عملکرد چند سلولی از طریق شبکه های بافت چند لایه” معرفی شده است:
Predicting Multicellular Function through Multi-layer Tissue Networks
این مجموعه داده، شامل مجموعه های ژن موقعیتی، مجموعه های ژن الگو و امضای ایمنی به عنوان ویژگی های هر گره یا راس است (50 ویژگی در مجموع)
به انگلیسی به ترتیب
gene sets, motif gene sets and immunological signatures
و مجموعه های انطباق ژنی(gene ontology sets) به عنوان برچسب یا لیبل (در مجموع 121) است.
این مجموعه داده به طور عمومی برای ارزیابی کارایی شبکههای عصبی گراف (GNNs) در کارهای طبقهبندی گراف استفاده میشود.
در PyTorch Geometric، میتوان با استفاده از کلاس torch_geometric.datasets.PPI، مجموعه داده PPI را بارگیری کرده و پیشپردازش کرد.
import os.path as osp
from torch_geometric.datasets import PPI
path = osp.join('.', 'data', 'PPI')
train_dataset = PPI(path, split='train')
val_dataset = PPI(path, split='val')
test_dataset = PPI(path, split='test')
آمار این دیتاست:
|
#graphs |
#nodes |
#edges |
#features |
#tasks |
|---|---|---|---|---|
| 20 |
~2,245.3 |
~61,318.4 |
50 |
121 |






دیدگاهتان را بنویسید